标注样例

例句“A C-to-A transversion in DAX1 gene resulted in a severe loss of DAX1 repressor activity, thus resulted in loss of leydig cell responsiveness”中: 任务一:实体标注一共有七处: 1、“C-to-A transversion”标注为“Var”; 2、“DAX1 gene”标注为“Gene”; 3、“loss”标注为“NegReg”; 4、“DAX1”标注为“Gene”; 5、“repressor activity”标注为MPA; 6、“loss”标注为“NegReg”; 7、“leydig cell responsiveness”标注为CPA; 任务二:语义角色的识别有6个: 1、由“DAX1 gene”指向“C-to-A transversion”的语义角色关系是“ThemeOf”,因为发生该突变的主体是“DAX1 gene” 2、由“C-to-A transversion”指向第一个“loss”的语义角色关系是“CauseOf”,因为突变发生后导致了DAX1基因抑制活动的丧失 3、由“DAX1”指向“repressor activity”的语义角色关系是“ThemeOf”,因为发生该抑制活性的主体是“DAX1”基因 4、由“repressor activity” 指向第一个“loss”的语义角色关系是“ThemeOf”,因为DAX1基因抑制活动的丧失事件的主体是抑制活动“repressor activity” 5、由“leydig cell responsiveness”指向第二个“loss”的语义角色关系是“ThemeOf”,因为间质细胞反应性的丧失事件的主体是“leydig cell responsiveness” 6、由第一个“loss”指向第二个“loss”的语义角色关系是“CauseOf”,因为DAX1基因抑制活动的丧失导致了间质细胞反应性的丧失
任务三: 例句1:Mutations in SHP-2 phosphatase that cause hyperactivation of its catalytic activity have been identified in human leukemias, particularly juvenile myelomonocytic leukemia. 从生物学的观点来看,催化活性的过度激活(hyperactivation of its catalytic activity)显然是功能获得的一种描述。因此,这句话承载着明确的语义信息,即基因“SHP-2”在突变后发挥着与“少年粒细胞白血病”( juvenile myelomonocytic leukemia)相关的“GOF”功能。因此任务三从这个句子中提取出的三元组就是“SHP-2; GOF; juvenile myelomonocytic leukemia ” 例句2:Lynch syndrome (LS) caused by mutations in DNA mismatch repair genes MLH1. 这句话描述了疾病“Lynch syndrome”与基因“MLH1”之间的关联,但短语“caused by”意味着没有损失或获得,因此这句话中的三元组应该是“MLH1; REG; Lynch syndrome”。 例句3:Here, we describe a fourth case of a human with a de novo KCNJ6 (GIRK2) mutation, who presented with clinical findings of severe hyperkinetic movement disorder and developmental delay. Heterologous expression of the mutant GIRK2 channel alone produced an aberrant basal inward current that lacked G protein activation, lost K+ selectivity and gained Ca2+ permeability. “失去K+选择性,获得Ca2+渗透性”( lost K+ selectivity and gained Ca2+ permeability)的描述同时显示了LOF和GOF,因此功能变化不能标记为LOF或GOF,而是标记为COM,因此这句话中的三元组应该是“GIRK2; COM; hyperkinetic movement disorder”
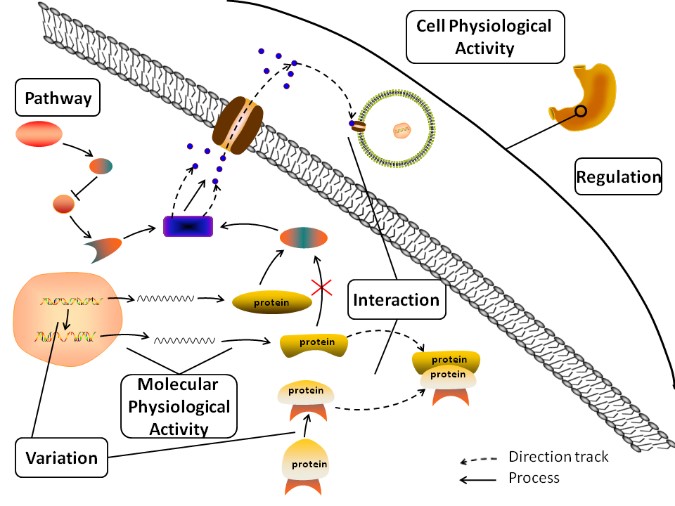 AGAC语料库设计了5个语义触发标签和3个根调控触发标签作为分子水平和细胞水平的生物实体标注,重点关注基因突变导致的生物过程中功能变化的信息,上图说明了对生物活动给不同标注的依据。
更具体的标注指南说明见:
Yuxing Wang, Xinzhi Yao, Kaiyin Zhou, Xuan Qin, Jin-Dong Kim, Kevin Bretonnel Cohen, Jingbo Xia*. Guideline Design of an Active Gene Annotation Corpus for the Purpose of Drug Repurposing. 2018 11th International Congress on Image and Signal Processing, BioMedical Engineering and Informatics (CISP-BMEI 2018), pp:1-5, Oct, 2018, Beijing
AGAC语料库设计了5个语义触发标签和3个根调控触发标签作为分子水平和细胞水平的生物实体标注,重点关注基因突变导致的生物过程中功能变化的信息,上图说明了对生物活动给不同标注的依据。
更具体的标注指南说明见:
Yuxing Wang, Xinzhi Yao, Kaiyin Zhou, Xuan Qin, Jin-Dong Kim, Kevin Bretonnel Cohen, Jingbo Xia*. Guideline Design of an Active Gene Annotation Corpus for the Purpose of Drug Repurposing. 2018 11th International Congress on Image and Signal Processing, BioMedical Engineering and Informatics (CISP-BMEI 2018), pp:1-5, Oct, 2018, Beijing
 上图是“地塞米松”药物发现过程的流程图,通过文本挖掘完全发现了突变DAX1的LOF功能变化,且没有任何给定药物的先验信息,任何针对DAX1的匹配药物的发现都是有价值的。为了验证这一点,于是在DrugBank中搜索DAX1的靶向信息,发现了一种名为“地塞米松”的药物,它不仅是一种激动剂,而且靶向DAX1。
更多AGAC在抗癫痫药物中的应用见已发表文献:
Yuxing Wang, Kaiyin Zhou, Jin-Dong Kim, Kevin Cohen, Mina Gachloo, Yuxin Ren, Shanghui Nie, Xuan Qin, Panzhong Lu, Jingbo Xia*. An Active Gene Annotation Corpus and Its Application on Anti-epilepsy Drug Discovery. BIBM 2019: International Conference on Bioinformatics & Biomedicine. Page: 512-519, San Diego, U.S, Nov, 2019.
4、更多AGAC相关信息请点击门户页面:
上图是“地塞米松”药物发现过程的流程图,通过文本挖掘完全发现了突变DAX1的LOF功能变化,且没有任何给定药物的先验信息,任何针对DAX1的匹配药物的发现都是有价值的。为了验证这一点,于是在DrugBank中搜索DAX1的靶向信息,发现了一种名为“地塞米松”的药物,它不仅是一种激动剂,而且靶向DAX1。
更多AGAC在抗癫痫药物中的应用见已发表文献:
Yuxing Wang, Kaiyin Zhou, Jin-Dong Kim, Kevin Cohen, Mina Gachloo, Yuxin Ren, Shanghui Nie, Xuan Qin, Panzhong Lu, Jingbo Xia*. An Active Gene Annotation Corpus and Its Application on Anti-epilepsy Drug Discovery. BIBM 2019: International Conference on Bioinformatics & Biomedicine. Page: 512-519, San Diego, U.S, Nov, 2019.
4、更多AGAC相关信息请点击门户页面: